HIKESHI - HIKESHI
| HIKESHI | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| |||||||||||||||||||||||||
| Tanımlayıcılar | |||||||||||||||||||||||||
| Takma adlar | HIKESHI, HSPC179, Hikeshi, L7RN6, OPI10, HSPC138, C11orf73, HLD13, kromozom 11 açık okuma çerçevesi 73, Hikeshi, ısı şoku proteini nükleer ithalat faktörü, ısı şoku proteini nükleer ithalat faktörü hikeshi | ||||||||||||||||||||||||
| Harici kimlikler | OMIM: 614908 MGI: 96738 HomoloGene: 6908 GeneCard'lar: HIKESHI | ||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| Ortologlar | |||||||||||||||||||||||||
| Türler | İnsan | Fare | |||||||||||||||||||||||
| Entrez | |||||||||||||||||||||||||
| Topluluk | |||||||||||||||||||||||||
| UniProt | |||||||||||||||||||||||||
| RefSeq (mRNA) | |||||||||||||||||||||||||
| RefSeq (protein) | |||||||||||||||||||||||||
| Konum (UCSC) | Chr 11: 86.3 - 86.35 Mb | Chr 7: 89.92 - 89.94 Mb | |||||||||||||||||||||||
| PubMed arama | [3] | [4] | |||||||||||||||||||||||
| Vikiveri | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
HIKESHI bir protein akciğer ve çok hücreli organizma gelişiminde önemli[5] bu, insanlarda HIKESHI gen.[6] HIKESHI, insanlarda kromozom 11'de ve farelerde kromozom 7'de bulunur. Benzer diziler (ortologlar ) çoğu hayvan ve mantar türünde bulunur. Fare homologu, 7. kromozomdaki öldürücü gen Rinchik 6 proteini tarafından kodlanmıştır l7Rn6 gen.[7] Farelerde l7Rn6 proteini bozulduğunda, fareler şiddetli amfizem doğumda düzensizliğin bir sonucu olarak Golgi cihazı ve içinde anormal veziküler yapıların oluşumu kulüp hücreleri.[5][güncellenmesi gerekiyor ]
Gen
HIKESHI, Homo sapiens'te protein kodlayan bir gendir. Gen için alternatif isimler FLJ43020, HSPC138, HSPC179 ve L7RN6'dır. Q14.2 alanındaki kromozom 11'in uzun kolu üzerinde bulunan, intronlar ve Eksonlar artı iplikçiğin 42.698 baz çiftidir. mRNA HIKESHI Varyantı 1, kodlama bölgelerini temsil eden 239 ila 832 baz çiftleri ile 1.183 baz çiftine karşılık gelen 1, 3, 4, 5 ve 7 eksonlarını içerir.
Alternatif Ekleme
Varyant 1, en uzun ve en yaygın protein kodlama varyantıdır. Diğer üç ana varyant, okuma çerçevesini atarak mRNA dizisinin erken sonlandırılmasına neden olan ve protein bozunmasına neden olan alternatif bir ekson dizisi kullanır. Aşağıdaki tablo, farklı varyantları ve ekson kullanımını göstermektedir.
| Varyant | Ekson 1 | Ekson 2 | Ekson 3 | Ekson 4 | Ekson 5 | Ekson 6 | Ekson 7 | Protein Kodlaması |
|---|---|---|---|---|---|---|---|---|
| 1 | x | x | x | x | x | Evet | ||
| 2 | x | x | x | x | x | x | Hayır | |
| 3 | x | x | x | x | x | Hayır | ||
| 4 | x | x | x | x | Hayır |
Yukarıdaki tabloda gösterilen dört değişken en yaygın olanlardır izoformlar insan hücrelerinde bulunur. İki alternatif promoter kullanan toplam 13 alternatif olarak eklenmiş sekans ve üç eklenmemiş form vardır. MRNA varyantları, 8 farklı ekson, alternatif, üst üste binen eksonların kombinasyonu ve intronlar. Alternatifin yanı sıra ekleme mRNA'lar 3'ün ucunda kesilerek farklılık gösterir. Varyant 1, bir proteini kodladığı gösterilen on mRNA'dan biridir, geri kalanı ise anlamsız aracılı mRNA bozunmasına bağlı görünmektedir.[8] C11orf73 izoformlarının gösterimi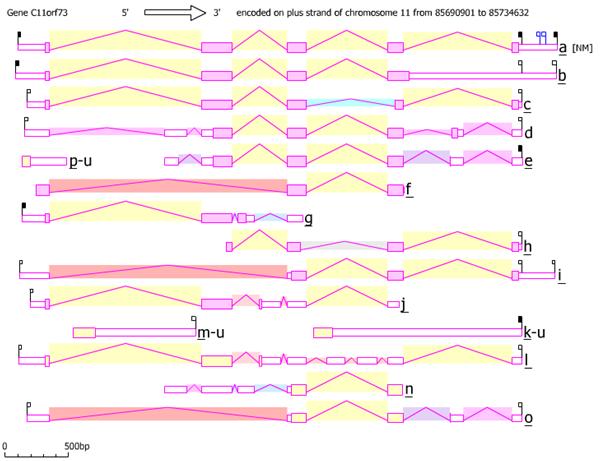
Organizatör
Promoter bölgesi, GXP 47146, ElDorado kullanılarak bulundu[9] Genomatix'ten araç. 840 bp sekansı, 86012753 ila 86013592 arasındaki DNA noktalarında HIKESHI geninden önce yer almaktadır. Promoter, 12 ortologdan 12'sinde korunur ve 6 ilgili transkript için kodlar.
Genomatix ElDorado aracından korunan transkripsiyon faktörü bağlama siteleri:
| Ayrıntılı Aile Bilgileri | Nereden | İçin | Çapa | Oryantasyon | Korunan Mus musculus | Matrix Sim | Sıra | Oluşum |
|---|---|---|---|---|---|---|---|---|
| Hücre döngüsü düzenleyicileri: Hücre döngüsü homoloji öğesi | 137 | 149 | 143 | + iplik | korunmuş | 0.943 | ggacTTGAattca | 1 |
| GATA bağlayıcı faktörler | 172 | 184 | 178 | + iplik | korunmuş | 0.946 | taaAGATttgagg | 1 |
| Omurgalı TATA bağlayıcı protein faktörü | 193 | 209 | 201 | + iplik | korunmuş | 0.983 | tcctaTAAAatttggat | 1 |
| Isı şok faktörleri | 291 | 315 | 303 | + iplik | korunmuş | 0.992 | cacagaaacgttAGAAgcatctctt | 4 |
| İnsan ve kemirgen ETS1 faktörleri | 512 | 532 | 522 | + iplik | korunmuş | 0.984 | taagccccGGAAgtacttgtt | 3 |
| Çinko parmak transkripsiyon faktörü RU49, Zipro1 | 522 | 528 | 525 | + iplik | korunmuş | 0.989 | aAGTAct | 2 |
| Krueppel gibi transkripsiyon faktörleri | 618 | 634 | 626 | + iplik | korunmuş | 0.925 | tggaGGGGcagacaccc | 1 |
| SOX / SRY-cinsiyet / testis belirleme ve HMG kutu faktörleri | 636 | 658 | 647 | + iplik | korunmuş | 0.925 | cccgcaAATTctggaaggttctt | 1 |

Sonlandırma
MRNA ürününün sonlandırılması, genin cDNA'sı içinde kodlanır. Bir mRNA ürününün sonlandırılması genellikle üç ana özelliğe sahiptir: poli A sinyali, poli bir kuyruk ve bir dizi alanı oluşturabilen bir gövde halkası yapı. Poli A sinyali, altı nükleotit uzunluğunda bir dizidir ve oldukça korunmuş bir bölgedir. Ökaryotlarda dizi, AATAAA'dır ve poli A bölgesinden yaklaşık 10-30 nükleotit içinde bulunur. AATAAA dizisi, sinyal dizisinden sonra 10-30 baz çiftinin mRNA ürününün poliadenilasyonunu işaret eden yüksek oranda korunmuş, ökaryotik bir poliA sinyalidir. C11orf73 için polyA sitesi GTA'dır.
Gen ifadesi
HIKESHI'nin ortalamanın 2,3 katı üzerinde yüksek bir seviyede her yerde ifade edildiği belirlendi. C11orf73, çok sayıda insan dokusunda ifade edilir.[10][11] UniGene üzerindeki İfade Profilleri ve EST Profili arasında, büyük olasılıkla dokudaki küçük numune boyutları nedeniyle yalnızca 11 dokunun C11orf73'ü ifade etmediği gösterildi.
Protein
İnsan HIKESHI geni adı verilen bir proteini kodlar karakterize edilmemiş protein C11orf73.[6] Homolog fare L7rn6 geni, 7. kromozomda öldürücü gen Rinchik 6.[7]
1 mfgclvagrl vqtaaqqvae dkfvfdlpdy esinhvvvfm lgtipfpegm ggsvyfsypd61 sngmpvwqll gfvtngkpsa eğer
Kodlanmış insan proteini, 197 amino asit uzunluğunda ve 21.628 Dalton ağırlığındadır. Fare proteinine benzer şekilde, insan HIKESHI proteininin varsayımsal işlevi, akciğer hücrelerindeki salgılama aparatının organizasyonu ve işlevidir.[5]
| Bilinmeyen işlevli protein (DUF775) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Tanımlayıcılar | |||||||||
| Sembol | DUF775 | ||||||||
| Pfam | PF05603 | ||||||||
| InterPro | IPR008493 | ||||||||
| |||||||||
protein alanı DUF775 olarak bilinen (Bilinmeyen Fonksiyon 775 Alanı), hem insan HIKESHI hem de fare L7rn6 proteinlerinde bulunur. DUF775 alanı, protein ile aynı uzunlukta, 197 amino asit uzunluğundadır. Tanım gereği DUF 775 süper ailesini oluşturan diğer proteinler C11orf73'ün tüm ortologlarını içerir.
Hidropati analizi, proteinde geniş bir hidrofobik bölge olmadığını gösterir ve dolayısıyla HIKESHI'nin sitoplazmik bir protein olduğu sonucuna varılır. C11orf73'ün izoelektrik noktası 5.108 olup, daha asidik bir ortamda en iyi şekilde çalıştığını göstermektedir.

SNP
Tek SNP,[13] veya tek nükleotid polimorfizmi C11orf73 dizisi için, protein içinde bir amino asit değişikliğine neden olur. Diğer SNP'lerin eksikliği büyük olasılıkla HIKESHI'nin yüksek düzeyde korunmasından ve proteindeki bir mutasyonun organizmaya verdiği ölümcül etkiden kaynaklanmaktadır. SNP için fenotip bilinmemektedir.
| Fonksiyon | dbSNP Aleli | Protein Kalıntısı | Codon Konumu | Amino Asit Konumu |
|---|---|---|---|---|
| Referans | C | Proline [P] | 1 | 47 |
| Yanlış anlam | G | Alanin [A] | 1 | 47 |
Gene Mahalle
HIKESHI'yi çevreleyen genler CCDC81'dir, ME3, ve EED. Genetik mahalleye, çevreleyen genlerin işlevine bakılarak genin olası işlevini daha iyi anlamak için bakılır.

CCDC81 geni, karakterize edilmemiş bir protein ürününü kodlar ve artı şerit üzerinde yönlendirilir. CCDC81, 81 izoform 1 içeren sarmal sargılı etki alanına yöneliktir.
ME3 geni, mitokondriyal malik enzim 3 öncüsü anlamına gelir. Malik enzim, oksidatif dekarboksilasyonunu katalize eder. malate -e piruvat ikisinden birini kullanarak NAD + veya bir kofaktör olarak NADP +. Memeli dokuları, malik enzimin 3 farklı izoformunu içerir: bir sitosolik NADP (+) bağımlı izoform, mitokondriyal NADP (+) bağımlı izoform ve mitokondriyal NAD (+) bağımlı izoform. Bu gen, mitokondriyal NADP (+) bağımlı izoformu kodlar. Bu gen için birden fazla alternatif olarak eklenmiş transkript varyantı bulunmuştur, ancak bazı varyantların biyolojik geçerliliği belirlenmemiştir.[15]
EED geni, embriyonik ektoderm geliştirme izoformu b ve bir üyesidir Polycomb grubu (PcG) ailesi. PcG ailesi üyeleri, ardışık hücre nesilleri boyunca genlerin transkripsiyonel baskılayıcı durumunun korunmasında rol oynayan multimerik protein kompleksleri oluşturur. Bu protein, sitoplazmik kuyruğu olan zeste 2'nin geliştiricisi ile integrin beta7, immün yetmezlik virüsü tip 1 (HIV -1) MA proteini ve histon deasetilaz proteinleri. Bu protein, histon deasetilasyon yoluyla gen aktivitesinin bastırılmasına aracılık eder ve integrin fonksiyonunun spesifik bir düzenleyicisi olarak hareket edebilir. Bu gen için farklı izoformları kodlayan iki transkript varyantı tanımlanmıştır.[16]
Etkileşimler
STRING programları[17] ve Sigma-Aldrich’in Favori Gen[18] C11orf73 ile olası protein etkileşimlerini önerdi. ARGUL1, CRHBP ve EED, metin madenciliğinden ve HNF4A, Sigma-Aldrich'ten elde edildi.
| Protein | Açıklama | Yöntem | Puan |
|---|---|---|---|
| ARGUL1 | Bilinmeyen | Metin madenciliği | 0.712 |
| CRHBP | Kortikotropin salgılayan hormon bağlayıcı protein | Metin madenciliği | 0.653 |
| EED | Embriyonik ektoderm gelişimi | Metin madenciliği | 0.420 |
| HNF4A | Transkripsiyon düzenleyici | Sigma-Aldrich | Yok |
ARGUL1, işlevi bilinmeyen, bilinmeyen bir proteindir. CRHBP HIKESHI'yi içeren veya aktive eden bir sinyal kademesinde muhtemelen rol oynayabilen kortikotrofin salgılayan hormon bağlayıcı bir proteindir. C11orf73'ün komşu bir proteini olan EED, bir embriyonik ektoderm geliştirme proteinidir ve Polycomb-group (PcG) ailesinin bir üyesidir. PcG ailesi üyeleri, ardışık hücre nesilleri boyunca genlerin transkripsiyonel baskılayıcı durumunun korunmasında rol oynayan multimerik protein kompleksleri oluşturur. HNF4A bir transkripsiyon düzenleyicidir ve HNF4A'nın C11orf73'ün ekspresyonunu düzenleyip düzenlemediği veya basitçe onunla etkileşime girip girmediği bilinmemektedir.[12
Evrimsel Tarih

Organizmaların evrimsel geçmişi, filogenetik bir ağaç oluşturmak için zaman referansları olarak ortolog dizileri kullanılarak belirlenebilir. CLUSTALW[19] çoklu dizileri karşılaştırdığında, program C11orf73 ortologlarına dayanan böyle bir filogenetik ağaç oluşturmak için de kullanılabilir. Sağdaki ağaç, üretilen filogenetik ağacı, sapma zamanına dayalı bir zaman çizgisiyle gösterir. HIKESHI ortologlarından yapılan ağaç, literatürdeki filogenetik ağaçla aynıdır, hatta balıklar, kuşlar ve mantarlar gibi benzer organizmaları bir arada gruplandırır.
Ortologlar
Homolog diziler, bir türleşme olayı ile ayrılmışlarsa ortologdur: Bir tür iki ayrı türe ayrıldığında, ortaya çıkan türlerdeki tek bir genin farklı kopyalarının ortolog olduğu söylenir. Ortologlar veya ortolog genler, ortak bir atadan geldikleri için birbirine benzeyen farklı türlerdeki genlerdir. Ortolog diziler, taksonomik sınıflandırmada ve organizmaların filogenetik çalışmalarında yararlı bilgiler sağlar. Genetik ıraksama modeli, organizmaların ilişkisini izlemek için kullanılabilir. Çok yakından ilişkili iki organizmanın, iki ortolog arasında çok benzer DNA dizileri göstermesi muhtemeldir. Tersine, başka bir organizmadan evrimsel olarak daha da uzaklaştırılan bir organizmanın, incelenen ortologların sıralamasında daha büyük bir sapma göstermesi muhtemeldir.
Kromozom Tablo 11 açık okuma çerçevesi 73 Ortologlar
| Türler | Yaygın isim | Protein Adı | Erişim numarası | NT Uzunluğu | NT Kimliği | AA Uzunluğu | AA Kimliği | E-Değeri |
|---|---|---|---|---|---|---|---|---|
| Homo sapiens | İnsan | C11orf73 | NM_016401 | 1187 bp | 100% | 197 aa | 100% | 0 |
| Bon taurus | İnek | LOC504867 | NP_001029398 | 996 bp | 73.60% | 197 aa | 98% | 5.30E-84 |
| Mus musculus | Fare | l7Rn6 | NP_080580 | 1045 bp | 72.90% | 197 aa | 97% | 4.80E-83 |
| Gallus gallus | Tavuk | LOC427034 | Yok | 851 bp | 56.20% | 197 aa | 88.3% | 5.60E-76 |
| Taeniopygia guttata | Zebra fincanı | LOC100190155 | ACH44077 | 997 bp | 61.60% | 997 aa | 87.80% | 1.20E-75 |
| Xenopus laevis | Kurbağa | MGC80709 | NP_001087012 | 2037 bp | 36.50% | 197 aa | 86.80% | 1.70E-75 |
| Oncorhynchus mykiss | Gökkuşağı alabalığı | CK073 | NP_001158574 | 940 bp | 52.20% | 197 aa | 75.10% | 2.70E-66 |
| Tetradon nigroviridis | Tetradon | isimsiz protein ürünü | CAF89643 | Yok | Yok | 197 aa | 70.90% | 1.40E-61 |
| Trichoplax adhaerens | Trichoplax adhaerens | TRIADDRAFT_19969 | XP_002108733 | 600 bp | 33.10% | 199 aa | 52.30% | 2.00E-47 |
| Culex quinquefasciatus | Sivrisinek | korunmuş varsayımsal protein | XP_001843282 | 594 bp | 30.70% | 197 aa | 49.30% | 2.50E-41 |
| Drosophilia melanogaster | Uçmak | CG13926 | NP_647633 | 594 bp | 31.50% | 197 aa | 48.50% | 4.50E-39 |
| Laccaria bicolor | Mantar | tahmini protein | XP_001878996 | 696 bp | 36.40% | 202 aa | 35.20% | 8.30E-24 |
| Candida albicans | Mantarlar | CaO19.13758 | XP_716157 | 666 bp | 36.10% | 221 aa | 24% | 5.70E-11 |
Tablo, protein adı, erişim numaraları, nükleotid kimliği, protein kimliği ve E değerleri ile birlikte 13 sekansı (12 ortolog, 1 orijinal sekans) göstermektedir. Erişim numaraları, NCBI Protein veri tabanındaki kimlik numaralarıdır. Nükleotit sekansına, erişim numarasını veren ve nükleotidin sekans sayfasına bir bağlantı olan DBSOURCE'den protein sekans sayfasından erişilebilir. Her ortolog ve ilgili organizması için hem nükleotit hem de protein dizisinin uzunluğu da tabloda listelenmiştir. Dizi uzunluklarının yanında ortologun orijinal HIKESHI genine kimlikleri bulunur. Kimlikler ve E-değerleri, SDSC Biology Workbench'ten global hizalama programı ALIGN ve NCBI'den BLAST kullanılarak elde edildi.
Grafik, çoğunlukla doğrusal bir eğri oluşturmak için ortologun organizmanın sapma süresine karşı yüzde özdeşliğini gösterir. Eğri içindeki iki ana eklem, sırasıyla yaklaşık 450 milyon yıl ve 1150 milyon yıl önceki gen kopyalanması zamanlarını gösteriyor. Gen duplikasyonlarından elde edilen paraloglar muhtemelen HIKESHI'nin yüksek oranda korunmuş ortologlarından o kadar farklıdır ki Blink veya BLAST araçları kullanılarak bulunamamıştır.


N'nin düzeltilmiş değeri olan m değeri (100 amino asitlik bir segmentte meydana gelen toplam amino asit değişikliklerinin sayısı) (şablon diziden amino asit farklılıkları sayısı), λ (ortalama amino asit yıllık asit değişiklikleri, genellikle λE9 değerlerinde temsil edilir.
m / 100 = –ln (1-n / 100) λ = (m / 100) / (2 * T)


Referanslar
- ^ a b c GRCh38: Ensembl sürüm 89: ENSG00000149196 - Topluluk, Mayıs 2017
- ^ a b c GRCm38: Ensembl sürüm 89: ENSMUSG00000062797 - Topluluk, Mayıs 2017
- ^ "İnsan PubMed Referansı:". Ulusal Biyoteknoloji Bilgi Merkezi, ABD Ulusal Tıp Kütüphanesi.
- ^ "Mouse PubMed Referansı:". Ulusal Biyoteknoloji Bilgi Merkezi, ABD Ulusal Tıp Kütüphanesi.
- ^ a b c Fernández-Valdivia R, Zhang Y, Pai S, Metzker ML, Schumacher A (Ocak 2006). "l7Rn6, Fare Akciğer Gelişiminde Clara Hücre İşlevi için Gereken Yeni Bir Proteini Kodlar". Genetik. 172 (1): 389–99. doi:10.1534 / genetik.105.048736. PMC 1456166. PMID 16157679.
- ^ a b Zhang QH, Ye M, Wu XY, Ren SX, Zhao M, Zhao CJ, Fu G, Shen Y, Fan HY, Lu G, Zhong M, Xu XR, Han ZG, Zhang JW, Tao J, Huang QH, Zhou J , Hu GX, Gu J, Chen SJ, Chen Z (Ekim 2000). "CD34 + Hematopoietik Kök / Progenitör Hücrelerde İfade Edilen Daha Önce Tanımlanmamış 300 Gen için Açık Okuma Çerçeveli cDNA'ların Klonlanması ve Fonksiyonel Analizi". Genom Res. 10 (10): 1546–60. doi:10.1101 / gr.140200. PMC 310934. PMID 11042152.
- ^ a b Rinchik EM, Carpenter DA (1993). "N-etil-N-nitrosoüre ile indüklenen prenatal olarak ölümcül mutasyonlar, fare kromozomu 7'de embriyonik ektoderm gelişimi (eed) lokusu içinde en az iki tamamlama grubunu tanımlar". Anne. Genetik şifre. 4 (7): 349–53. doi:10.1007 / BF00360583. PMID 8358168. S2CID 24689449.
- ^ AceView NCBI Gen Bilgileri AceView Arşivlendi 7 Nisan 2006, WebCite
- ^ Promoter analizi için Genomatix ElDorade aracı ElDorado Ürün Sayfası Arşivlendi 6 Ekim 2008, Wayback Makinesi
- ^ "C11orf73 için ifade profili". GeneNote sürüm 2.4. Weizmann Bilim Enstitüsü. Eylül 2009.
- ^ "EST Profili - Hs.283322". Ulusal Biyoteknoloji Bilgi Merkezi, Birleşik Devletler Ulusal Tıp Kütüphanesi.
- ^ Saier Lab Biyoinformatik Grubu http://www.tcdb.org/progs/hydro.php
- ^ NCBI SNP Veritabanı https://www.ncbi.nlm.nih.gov/snp/ Arşivlendi 26 Ocak 2006, WebCite
- ^ NCBI Entrez https://www.ncbi.nlm.nih.gov/nuccore/NC_000011.9?from=86011067&to=86059171&report=graph
- ^ RefSeq NCBI Veritabanı https://www.ncbi.nlm.nih.gov/RefSeq/ Arşivlendi 30 Mart 2006, WebCite
- ^ "RefSeq: NCBI Referans Sırası Veritabanı".
- ^ STRING (Etkileşen Genlerin / Proteinlerin Alınması için Arama Aracı) http://string-db.org/ Arşivlendi 23 Temmuz 2010, WebCite
- ^ Sigma-Aldrich'in Favori Gen http://www.sigmaaldrich.com/life-science/your-favorite-gene-search.html
- ^ CLUSTALW Programı Julie D. Thompson, Desmond G. Higgins ve Toby J. Gibson http://workbench.sdsc.edu/ Arşivlendi 30 Mart 2006, WebCite
Dış bağlantılar
- İnsan C11orf73 genom konumu ve C11orf73 gen ayrıntıları sayfası UCSC Genom Tarayıcısı.




